U organizmów eukariotycznych sekwencja nukleotydów pierwotnego transkryptu (pre-mRNA) ulega wieloetapowej obróbce zwanej procesem dojrzewania. Zachodzi ona głównie w jądrze komórkowy i tylko poddane niej cząsteczki mogą przemieszczać się z jądra do cytoplazmy i uczestniczyć w przebiegu translacji. Obróbka pierwotnych transkryptów może służyć do regulacji ekspresji informacji genetycznej. Obecność intronów umożliwia bowiem wykorzystywanie alternatywnych miejsc składania genów oraz taksowanie i duplikację eksonów, przyczyniając się do powiększenia zasobów informacji genetycznej.
Składanie (ang. Splicing),
czyli precyzyjne wycinanie niekodujących intronów i łączenie pozostałych eksonów. Powstała sekwencja RNA zawiera ciągłą informację o strukturze budowanego białka i nazywana jest otwartą ramką odczytu (ang. open reading frame).
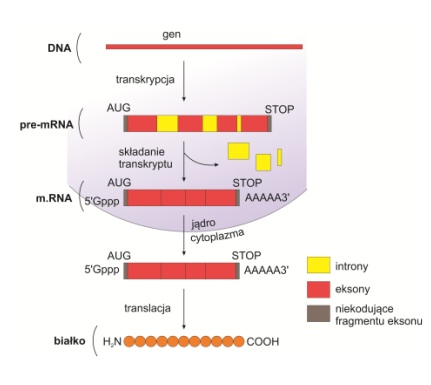
Rys. 1. Splicing
Introny mają różną długość i zaczynają się zwykle od sekwencji GU, a kończą na AG. Posiadają również ważny odcinek zwany miejscem rozgałęzienia (ang. branch site) biorący udział przy ich wycinaniu. Znajdujące się na stykach miejsca cięcia mają konserwatywną budowę, tzw. sekwencję konsensusu, która znacznie ułatwia ich identyfikację.

Rys. 2. Sekwencja konsensus
Istnieją różne mechanizmy składania transkryptu:
• samowycinanie – zachodzi m.in. genach mitochondrialnych; intron jest wycinany w sposób autokatalityczny, tzn. bez udziału białek;
• wycinanie z udziałem spliceosomów, czyli struktur złożonych z białek oraz czterech małocząsteczkowych RNA – snRNA (U1, U2, U5 i U4/U6). Przecięty przy końcu 5’ intron tworzy pętlę (produkt pośredni w kształcie lassa – ang. lariat intermediate). Dopiero potem przecinany jest koniec 3’, a całość uwalniana z pierwotnego transkryptu i trawiona enzymatycznie. Pozostałe eksony łączą się ze sobą.
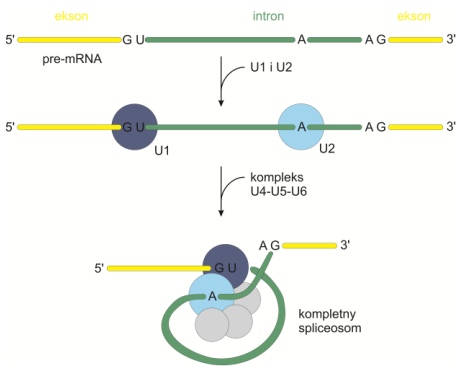
Rys. 3. Sposób składania spliceosomu
Redagowanie (ang. Editing)
Niektóre cząsteczki RNA podczas obróbki tracą lub zyskują nowe nukleotydy. Jest to jeden z mechanizmów regulacji ekspresji genów. Proces ten zachodzi w różnych częściach transkryptu, w kierunku od końca 3’ do 5’.W jego wyniku mogą powstawać nowe otwarte ramki odczytu, nowe miejsca startu translacji oraz sekwencje wyznaczające miejsce wycinania eksonów. U świdrowców bierze w nim udział korygujące RNA (ang. guide RNA, gRNA) spełniające rolę matrycy.
Modyfikacja cząsteczek mRNA
Zachodzi głównie w jądrze komórkowym i obejmuje oba końce nowopowstałego transkryptu:
• do końca 5’ doczepiana zostaje czapeczka (7-metyloguanozyna), która ułatwia transport cząsteczki z jądra do cytoplazmy, zabezpiecza transkrypt przed działaniem enzymów nukleolitycznych oraz uczestniczy przy wycinaniu intronów i inicjowaniu translacji;
• poliadenylacja końca 3’ , czyli przyłączenie ogona złożonego z poli(A) – nie zawierają go transkrypty genów kodujących białka histonowe. Rola tej modyfikacji nie jest do końca poznana, wiadomo jednak, że nie decyduje ona o pojawieniu się cząsteczki w cytoplazmie.
Usunięcie czapeczki i ogona poli(A) obniża trwałość i stabilność transkryptu.
Po stronie 3’ i 5’ cytoplazmatycznego mRNA występują dodatkowe niekodujące białek regiony. Biorą one udział w przekształcaniu RNA, transporcie cząsteczki, jej degradacji oraz translacji.
Modyfikacja cząsteczek tRNA
Transportowy RNA przepisywany jest w postaci dużych prekursorowych cząsteczek, zawierających więcej niż 1 tRNA. Ulegają one przekształceniom nukleolitycznym katalizowanym przez rybonukleazy. Podczas dalszych etapów zachodzi wycinanie intronów ulokowanych w pobliżu pętli antykodonowej, alkilacja nukleotydów oraz dołączenie charakterystycznej końcówki CCA przy końcu 3’. Jest ona odpowiedzialna za swoiste łączenie się z transportowanym aminokwasem.
Modyfikacja cząsteczek rRNA
U ssaków geny kodujące rRNA umiejscowione są na terenie jąderka. Pojedyncze cząsteczki silnie zmetylowanego, pierwotnego transkryptu 45S zawierają geny 18S, 5,8S i 28S rRNA i ulegają swoistym nukleolitycznym przekształceniom, podczas których degradowana jest ponad połowa oryginalnego pierwotnego transkryptu. Zachodzi również dalsza metyzacja oraz utworzenie podjednostek rybosomalnych, czyli połączenie rRNA z odpowiednimi białkami.
Autor: Anna Kurcek
Literatura:
• Lubert Stryer „Biochemia”, Wydawnictwo Naukowe PWN, Warszawa 2003;
• Robert K. Murray, Daryl K. Granner, Peter A. Mayes, Victor W. Rodwell “Biochemia Harpera” Wydanie III, Redaktor naukowy tłumaczenia Franciszek Kokot, Warszawa 1995, Wydawnictwo Lekarskie PZWL;
• Jerzy Bal :Biologia molekularna w medycynie. Elementy genetyki klinicznej”, Wydawnictwo Naukowe PWN, Warszawa 2001.

